import numpy as np
import pandas as pd
np.random.seed(123)
n = 100
data = pd.DataFrame({
'true': ['perro'] * 50 + ['gato'] * 50,
'probs': np.concatenate([
np.random.uniform(0.5, 1, 50),
np.random.uniform(0, 0.5, 50)
])
})
data['pred'] = data['probs'].apply(lambda x: 'perro' if x > 0.5 else 'gato')
indices_erreurs = np.random.choice(n, 15, replace=False)
data.loc[indices_erreurs, 'probs'] = 1 - data.loc[indices_erreurs, 'probs']
data.loc[indices_erreurs, 'pred'] = data.loc[indices_erreurs, 'pred'].map({'perro': 'gato', 'gato': 'perro'})Ejemplo de datos
Buscamos identificar los resultados de un modelo que trata de diferenciar predicciones de perros y gatos. Para evaluar su rendimiento, se utilizan métricas adaptadas que permiten medir cuán preciso, confiable y equilibrado es el modelo en sus predicciones.
set.seed(123)
n <- 100
data <- data.frame(
true = factor(c(rep("perro", 50), rep("gato", 50))),
probs = c(runif(50, 0.5, 1), runif(50, 0, 0.5))
)
data$pred <- factor(ifelse(data$probs > 0.5, "perro", "gato"))
indices_erreurs <- sample(1:n, 15)
data$probs[indices_erreurs] <- 1 - data$probs[indices_erreurs]
data$pred[indices_erreurs] <- ifelse(data$pred[indices_erreurs] == "perro", "gato", "perro") Métricas
Matriz de confusión
La matriz de confusión es una representación en tabla de las predicciones correctas e incorrectas:
\[ \begin{bmatrix} TP & FP \\ FN & TN \end{bmatrix} \]
- TP (True Positives) : Número de predicciones correctas para la clase positiva.
- FP (False Positives) : Número de predicciones incorrectas para la clase positiva.
- FN (False Negatives) : Número de predicciones incorrectas para la clase negativa.
- TN (True Negatives) : Número de predicciones correctas para la clase negativa.
from sklearn.metrics import confusion_matrix
import seaborn as sns
import matplotlib.pyplot as plt
conf_matrix = confusion_matrix(data['true'], data['pred'])
print("Matrice de confusion Python:")Matrice de confusion Python:print(conf_matrix)[[42 8]
[ 7 43]]plt.figure(figsize=(6, 4))
sns.heatmap(conf_matrix,
annot=True,
fmt='d',
xticklabels=['gato', 'perro'],
yticklabels=['gato', 'perro'])
plt.title('Matrice de confusion (Python)')
plt.xlabel('Valeur réelle')
plt.ylabel('Prédiction')
plt.show()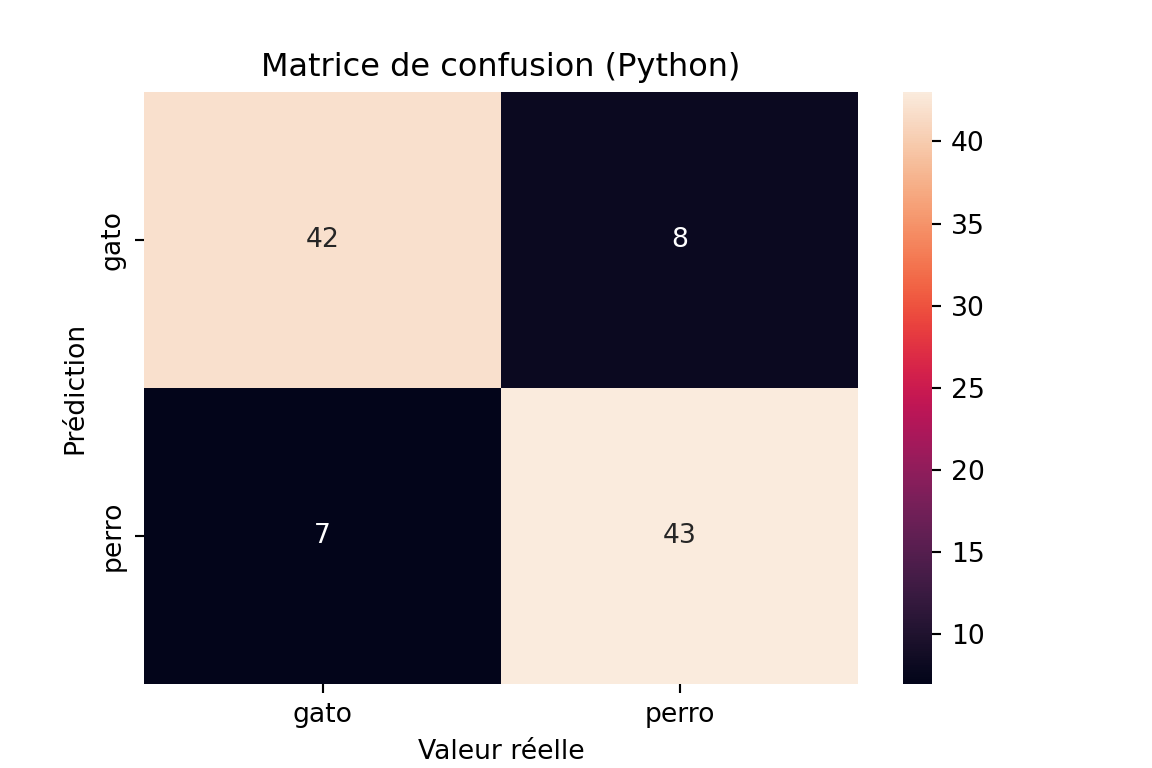
library(ggplot2)
print("Matrice de confusion R:")[1] "Matrice de confusion R:"table(Prédit = data$pred, Réel = data$true) Réel
Prédit gato perro
gato 44 9
perro 6 41conf_matrix_r <- as.data.frame(table(data$pred, data$true))
names(conf_matrix_r) <- c("Prediction", "Real", "Count")
ggplot(conf_matrix_r,
aes(x = Real, y = Prediction, fill = Count)) +
geom_tile() +
geom_text(aes(label = Count)) +
scale_fill_gradient(low = "white", high = "steelblue") +
theme_minimal() +
labs(title = "Matrice de confusion (R)")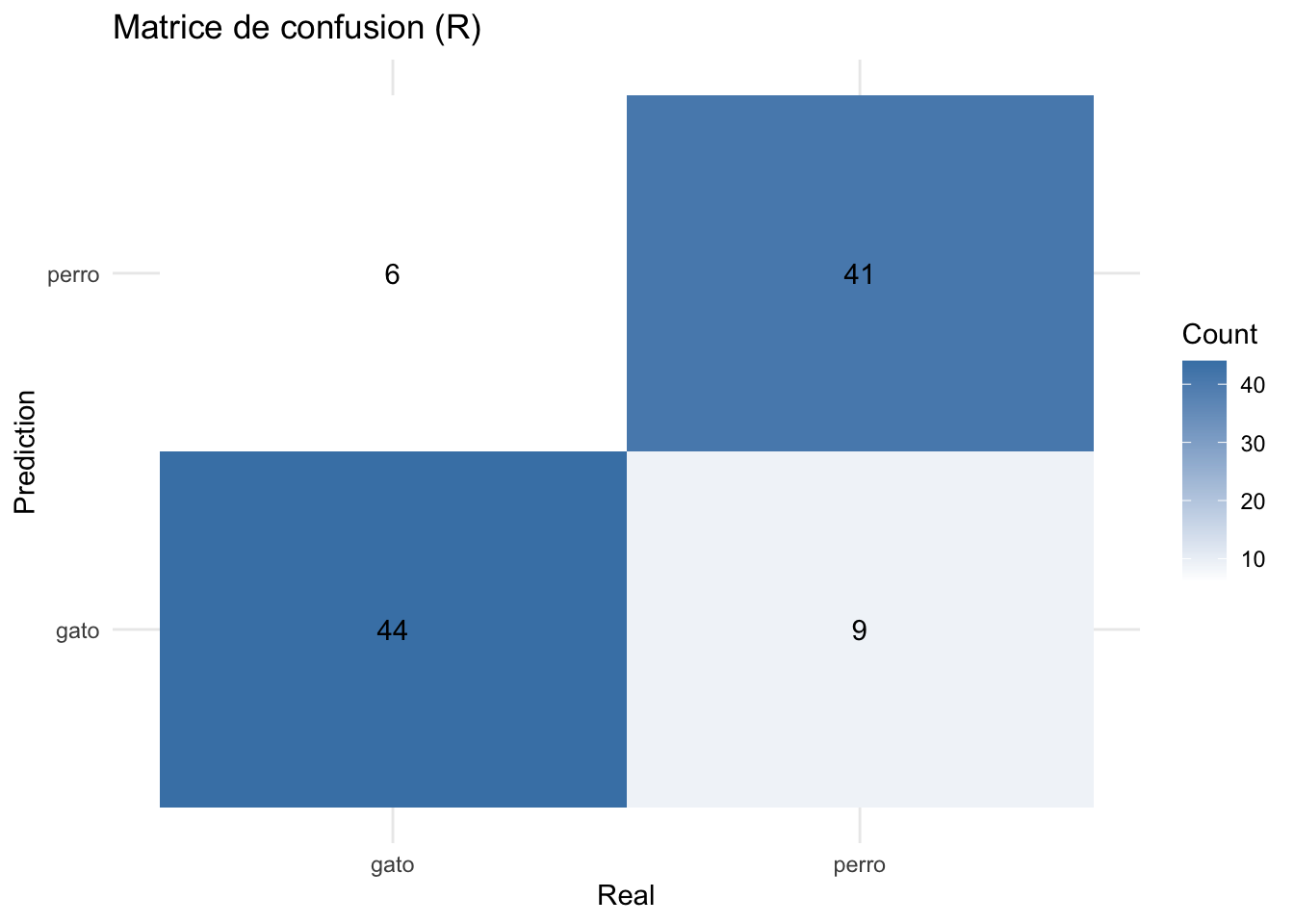
Exactitud (Accuracy)
La Exactitud mide la proporción de predicciones correctas entre el total de predicciones.
\[ \text{Exactitud} = \frac{\text{Verdaderos Positivos} + \text{Verdaderos Negativos}}{\text{Total}} \]
Ventajas : Fácil de entender e interpretar. Representa bien el rendimiento general de un modelo cuando las clases están equilibradas.
Desventajas : No tiene en cuenta el desequilibrio de clases. Un modelo puede tener una alta exactitud incluso si clasifica mal las clases minoritarias.
from sklearn.metrics import accuracy_score
acc_sklearn = accuracy_score(data['true'], data['pred'])
print(f"Accuracy (sklearn): {acc_sklearn:.3f}")Accuracy (sklearn): 0.850acc_simple <- mean(data$pred == data$true)
print(paste("Accuracy (simple):", round(acc_simple, 3)))[1] "Accuracy (simple): 0.85"Precisión
La precisión es la proporción de predicciones positivas correctas entre todas las predicciones positivas.
Ventajas : Útil cuando se quiere minimizar los falsos positivos. Por ejemplo, en escenarios donde una falsa alarma es costosa (como en la detección de fraudes).
Desventajas : No tiene en cuenta los falsos negativos, lo que puede ser problemático en ciertos casos, como cuando se quiere evitar los falsos negativos.
\[ \text{Précision} = \frac{\text{Verdaderos Positivos}}{\text{Verdaderos Positivos} + \text{Falsos Positivos}} \]
from sklearn.metrics import precision_score
precision_per_class = precision_score(
data['true'],
data['pred'],
average=None,
labels=['gato', 'perro']
)
print("recisión por clase (sklearn):")recisión por clase (sklearn):print(f"gato: {precision_per_class[0]:.3f}")gato: 0.857print(f"perro: {precision_per_class[1]:.3f}")perro: 0.843# Précision moyenne
precision_avg = precision_score(
data['true'],
data['pred'],
average='macro'
)
print(f"Precisión promedio: {precision_avg:.3f}")Precisión promedio: 0.850conf_matrix <- table(Prédit = data$pred, Réel = data$true)
precision_gato <- conf_matrix["gato","gato"] / sum(conf_matrix[,"gato"])
precision_perro <- conf_matrix["perro","perro"] / sum(conf_matrix[,"perro"])
print("\nrecisión por clase (manuel):")[1] "\nrecisión por clase (manuel):"print(paste("gato:", round(precision_gato, 3)))[1] "gato: 0.88"print(paste("perro:", round(precision_perro, 3)))[1] "perro: 0.82"precision_moy <- mean(c(precision_gato, precision_perro))
print(paste("Precisión promedio:", round(precision_moy, 3)))[1] "Precisión promedio: 0.85"Recall
El Recall mide la proporción de verdaderos positivos detectados entre todos los reales positivos. Es especialmente importante cuando se quiere minimizar los falsos negativos.
Ventajas : Útil en contextos donde es crucial capturar tantos casos positivos como sea posible (por ejemplo, en pruebas médicas).
Desventajas : Ignora los falsos positivos, lo que puede llevar a un aumento de los falsos positivos en ciertos casos.
\[ \text{Recall} = \frac{\text{Verdaderos Positivos}}{\text{Verdaderos Positivos} + \text{Falsos Negativos}} \]
from sklearn.metrics import recall_score
recall_per_class = recall_score(
data['true'],
data['pred'],
average=None,
labels=['gato', 'perro']
)
print("Recall por classe (sklearn):")Recall por classe (sklearn):print(f"gato: {recall_per_class[0]:.3f}")gato: 0.840print(f"perro: {recall_per_class[1]:.3f}")perro: 0.860# Recall moyen
recall_avg = recall_score(
data['true'],
data['pred'],
average='macro'
)
print(f"Recall promedio: {recall_avg:.3f}")Recall promedio: 0.850conf_matrix <- table(Prédit = data$pred, Réel = data$true)
recall_gato <- conf_matrix["gato","gato"] / sum(conf_matrix["gato",])
recall_perro <- conf_matrix["perro","perro"] / sum(conf_matrix["perro",])
print("\nRecall par classe (manuel):")[1] "\nRecall par classe (manuel):"print(paste("gato:", round(recall_gato, 3)))[1] "gato: 0.83"print(paste("perro:", round(recall_perro, 3)))[1] "perro: 0.872"# Recall moyen
recall_moy <- mean(c(recall_gato, recall_perro))
print(paste("Recall promedio:", round(recall_moy, 3)))[1] "Recall promedio: 0.851"F1-Score
El F1-Score es una medida combinada de la precisión y el recall (recuperación). Es la media armónica de ambas, y es útil cuando se desea equilibrar estas dos métricas. El F1-Score es especialmente útil cuando hay un desbalance entre las clases.
Ventajas : Toma en cuenta tanto los falsos positivos (FP) como los falsos negativos (FN), lo que lo hace útil en problemas con clases desbalanceadas.
Desventajas : Si la precisión o el recall son bajos, el F1-Score también será bajo.
\[ \text{F1-Score} = 2 \times\frac{\text{Précision} \times \text{Recall}}{\text{Précision} + \text{Recall}} \]
from sklearn.metrics import f1_score
# F1-score par classe
f1_per_class = f1_score(
data['true'],
data['pred'],
average=None,
labels=['gato', 'perro']
)
print("F1-Score par classe (sklearn):")F1-Score par classe (sklearn):print(f"gato: {f1_per_class[0]:.3f}")gato: 0.848print(f"perro: {f1_per_class[1]:.3f}")perro: 0.851# F1-score moyen
f1_avg = f1_score(
data['true'],
data['pred'],
average='macro'
)
print(f"F1-Score promedio: {f1_avg:.3f}")F1-Score promedio: 0.850precision_gato <- conf_matrix["gato", "gato"] / sum(conf_matrix[, "gato"])
recall_gato <- conf_matrix["gato", "gato"] / sum(conf_matrix["gato", ])
f1_gato <- 2 * (precision_gato * recall_gato) / (precision_gato + recall_gato)
precision_perro <- conf_matrix["perro", "perro"] / sum(conf_matrix[, "perro"])
recall_perro <- conf_matrix["perro", "perro"] / sum(conf_matrix["perro", ])
f1_perro <- 2 * (precision_perro * recall_perro) / (precision_perro + recall_perro)
print("\nF1-Score par classe (manuel):")[1] "\nF1-Score par classe (manuel):"print(paste("gato:", round(f1_gato, 3)))[1] "gato: 0.854"print(paste("perro:", round(f1_perro, 3)))[1] "perro: 0.845"# F1-score moyen
f1_moy <- mean(c(f1_gato, f1_perro))
print(paste("F1-Score moyen:", round(f1_moy, 3)))[1] "F1-Score moyen: 0.85"ROC-AUC (Area Under the Curve)
La curva ROC muestra la relación entre la tasa de verdaderos positivos (TPR) y la tasa de falsos positivos (FPR). El AUC (Área Bajo la Curva) mide la capacidad del modelo para diferenciar entre las clases.
Ventajas : Es eficaz para evaluar modelos con clases desbalanceadas, y permite comparar modelos con diferentes umbrales de clasificación.
Desventajas : A veces es difícil de interpretar en contextos muy específicos.
\[ AUC = \int_0^1 \text{TPR}(fpr) \, dfpr \]
from sklearn.metrics import roc_auc_score, roc_curve
roc_auc = roc_auc_score(data['true'] == 'perro', data['probs'])
print(f"ROC-AUC: {roc_auc:.3f}")ROC-AUC: 0.841fpr, tpr, thresholds = roc_curve(data['true'] == 'perro', data['probs'])
plt.figure(figsize=(8, 6))
plt.plot(fpr, tpr, color='blue', label=f'Curva ROC (AUC = {roc_auc:.2f})')
plt.plot([0, 1], [0, 1], color='gray', linestyle='--')
plt.xlabel('Tasa de Falsos Positivos (FPR)')
plt.ylabel('Tasa de Verdaderos Positivos (TPR)')
plt.title('Courbe ROC')
plt.legend(loc='lower right')
plt.show()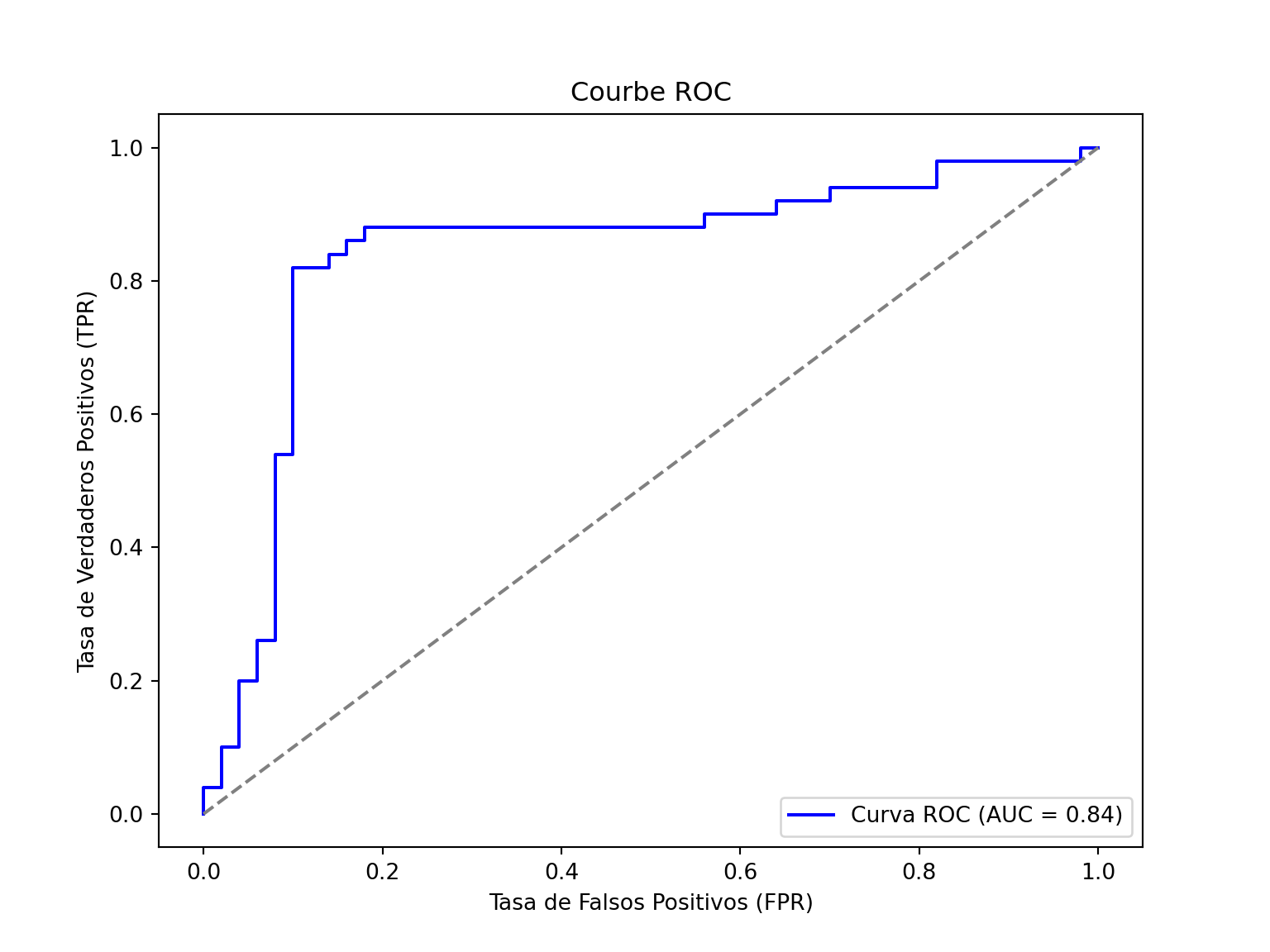
library(pROC)Type 'citation("pROC")' for a citation.
Attaching package: 'pROC'The following objects are masked from 'package:stats':
cov, smooth, varroc_auc <- roc(data$true == "perro", data$probs)$aucSetting levels: control = FALSE, case = TRUESetting direction: controls < casesprint(sprintf("ROC-AUC: %.3f\n", roc_auc))[1] "ROC-AUC: 0.856\n"roc_curve <- roc(data$true == "perro", data$probs)Setting levels: control = FALSE, case = TRUE
Setting direction: controls < casesplot(roc_curve, main = "Courbe ROC", col = "blue")
legend("bottomright", legend = paste("AUC =", round(roc_auc, 2)), col = "blue", lty = 1)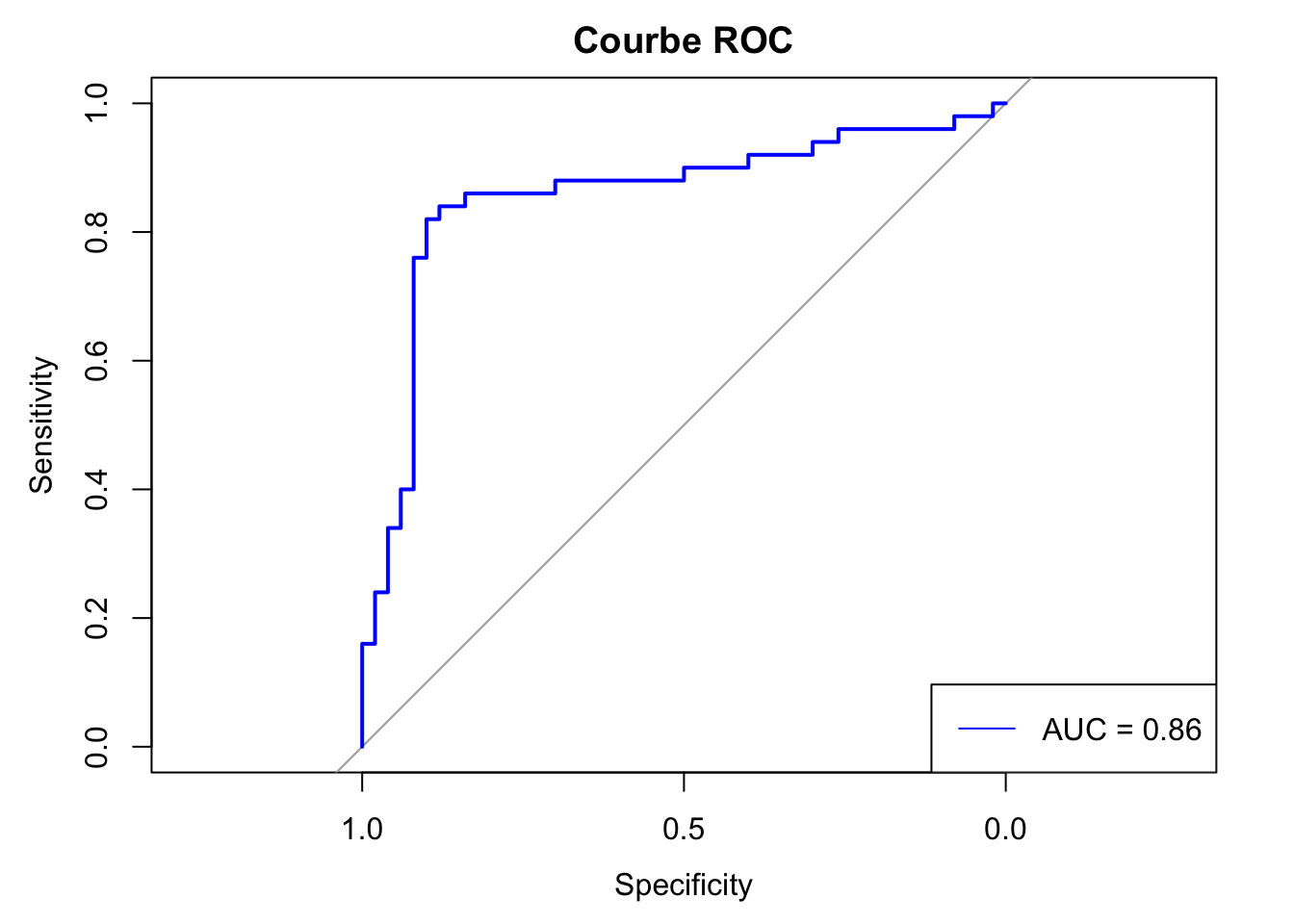
Log Loss (Logarithmic Loss)
El Log-Loss mide la calidad de las probabilidades asignadas por un modelo. Un valor de Log-Loss más bajo indica que el modelo asigna probabilidades más precisas.
Ventajas : Evalúa la confianza del modelo en sus predicciones, lo cual es importante para modelos que predicen probabilidades.
Desventajas : Es sensible a las predicciones incorrectas con alta confianza.
\[ \text{Log Loss} = - \frac{1}{N} \sum_{i=1}^{N} \left[ y_i \log(p_i) + (1 - y_i) \log(1 - p_i) \right] \]
from sklearn.metrics import log_loss
log_loss_value = log_loss(data['true'], np.vstack([1 - data['probs'], data['probs']]).T, labels=['gato', 'perro'])
print(f"Log-Loss: {log_loss_value:.3f}")Log-Loss: 0.520data$true_binary <- ifelse(data$true == "perro", 1, 0)
log_loss <- -mean(data$true_binary * log(data$probs) + (1 - data$true_binary) * log(1 - data$probs))
print(paste("Log Loss: ", round(log_loss, 4)))[1] "Log Loss: 0.4953"Matthews Correlation Coefficient (MCC)
El MCC mide la correlación entre las predicciones y los resultados reales, considerando los verdaderos positivos (TP), los falsos positivos (FP), los verdaderos negativos (TN) y los falsos negativos (FN). Es especialmente útil cuando las clases están desbalanceadas.
Ventajas : Proporciona un buen equilibrio entre todos los posibles errores (falsos positivos, falsos negativos, verdaderos positivos y verdaderos negativos).
Desventajas : Es más difícil de interpretar que métricas como la precisión o el F1-Score.
\[ MCC = \frac{TP \times TN - FP \times FN}{\sqrt{(TP + FP)(TP + FN)(TN + FP)(TN + FN)}} \]
from sklearn.metrics import matthews_corrcoef
mcc_value = matthews_corrcoef(data['true'], data['pred'])
print(f"Matthews Correlation Coefficient (MCC): {mcc_value:.3f}")Matthews Correlation Coefficient (MCC): 0.700library(mltools)
mcc_value <- mcc(data$pred, data$true)
print(sprintf("Matthews Correlation Coefficient (MCC): %.3f\n", mcc_value))[1] "Matthews Correlation Coefficient (MCC): 0.701\n"